AIM算法实时增强超分辨率显微镜图像
当尝试以纳米精度测量分子结构时,每一点噪音都会出现在数据中:有人走过显微镜、建筑物中的微小振动,甚至外面的交通。一种新的处理技术可以实时消除光学显微镜数据中的噪音,使科学家能够以比以前高 10 倍以上的精度跟踪单个分子。
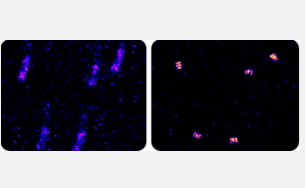
伊利诺伊大学香槟分校的一组生物工程研究人员推出了一种名为自适应交集最大化 (AIM) 的算法,该算法可以比标准方法更快地从超分辨率光学显微镜数据中去除高频噪声,从而获得更高的图像分辨率。该算法将使科学家能够比以前更轻松、更准确地研究化学和生物系统。这项研究发表在《科学进展》杂志上。
“起初,我们只是想开发一种快速算法,因为我们的实验室产生的数据太多,传统算法无法处理,但我们发现 AIM 还可以达到亚纳米精度,这在我们的领域是闻所未闻的,” 生物工程研究教授、这项研究的主要作者马红强说。“此外,它不需要像传统工具那样强大的计算能力。它可以在笔记本电脑上运行。我们希望让它成为所有显微镜用户的即插即用工具。”
近几十年来,单分子定位显微镜技术让科学家能够可视化分子级结构,突破了光学显微镜的根本限制。然而,在实践中,该技术受到不可控的噪声或“漂移”的限制,这些噪声会模糊图像,并阻止超分辨率显微镜达到最高分辨率。
“单分子定位实际上使用了一种相当简单的仪器,但真正影响图像分辨率的棘手部分是漂移,” 生物工程教授兼项目负责人杨刘说。“许多研究人员只消除低频漂移。消除高频漂移——由环境噪声引起的微小振动——需要大量计算,并且需要大量的时间和资源。”
消除漂移的标准方法基于图像帧之间的数学相关性。刘女士表示,她实验室的显微镜产生的图像数据量非常大,即使使用超级计算资源,图像相关性方法也需要数天时间。
AIM 也会比较相邻帧,但它会将每个数据点放在一个圆心(由定位精度定义)上,然后在其他帧中寻找该圆内的点。在“交点半径”内的重叠点被压缩为一个单一的定位。然后,对压缩后的点重复该过程一次。这些步骤占用的计算资源最少,而且比显微镜相机的采集时间更快。因此,可以实时生成漂移校正图像。
研究人员使用模拟数据和具有明确特征的 DNA 折纸结构对 AIM 进行了测试。该算法成功定位了这些结构,其精度小于 1 纳米,远高于标准图像相关方法(约 10 纳米)。
刘的实验室将把 AIM 整合到正在开发的用于增强疾病检测的高通量显微镜技术中。不过,刘还相信该算法将在整个生物学和生物工程领域得到应用。“这是一个快速且易于使用的工具,我们希望让整个社区都能广泛使用它,”她说。“我们正在使我们的软件公开可用。我们希望人们仅通过这一步后处理就能提高图像分辨率。”
免责声明:本答案或内容为用户上传,不代表本网观点。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。 如遇侵权请及时联系本站删除。
